研究背景
真核细胞在遭受外界不利条件的胁迫时,细胞稳态往往被扰乱,导致细胞功能的正常运行乃至存活都面临挑战。细胞中的内质网作为蛋白质合成、折叠、加工和运输的重要场所,外部刺激导致的蛋白质稳态失衡会引起未折叠或错误折叠蛋白质在内质网中的积累,引发内质网应激(ER stress)。在内质网(ER)应激时,内质网膜跨膜蛋白激酶/内切核酸酶肌醇需要酶1(IRE1)的激活是未折叠蛋白响应通路(UPR)的一个关键信号,激活的肌醇需要酶1(IRE1)可以形成大型聚集物/聚焦簇,但是其确切的结构特点、动态特征、功能特性迄今为止仍然大部分不明。
研究目的
为研究细胞抗逆应激的生物学机制,武汉大学生命科学学院刘勇教授课题组于2024年5月8日在国际学术期刊《Nature Cell Biology》(IF:21.3)上发表题为“Mammalian IRE1α dynamically and functionally coalesces with stress granules”的文章。
文章发现IRE1α能够在不同应激条件下与应激颗粒(SG)通过相分离形成共聚集簇,提示IRE1α与应激颗粒的协同共聚能够富集IRE1α-XBP1通路中的关键组分,为IRE1α蛋白机器提供了一个更为高效的运行平台,从而赋予细胞更强的应激处理能力(下图1)。
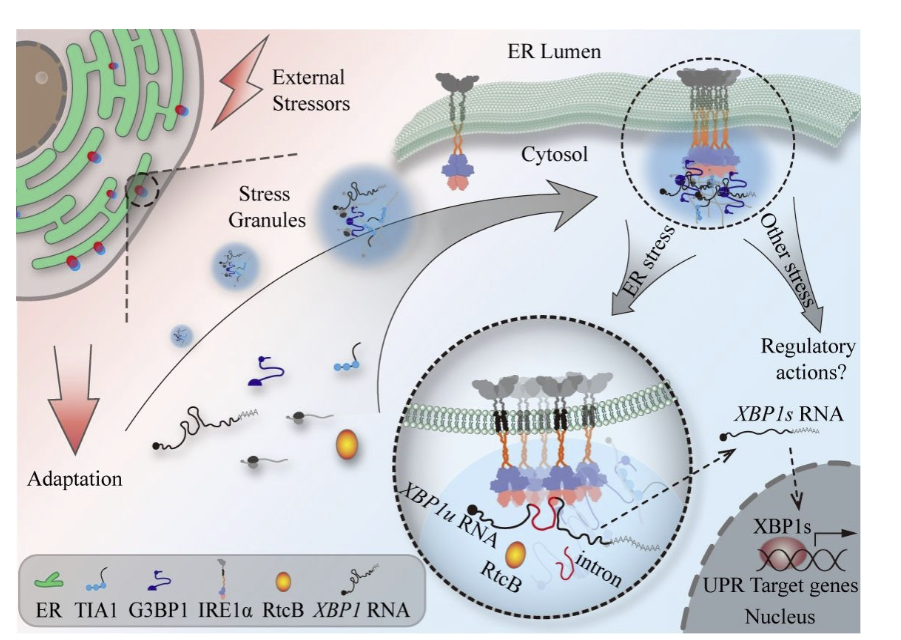
图1. 哺乳动物细胞中IRE1α-SG共聚集簇的功能机制示意图
(论文链接:https://doi.org/10.1038/s41556-
024-01418-7)
研究创新点
首次揭示未折叠蛋白响应分子IRE1α与应激颗粒共聚集的动态特征与功能机制,这些发现为理解细胞如何在内质网应激下调节IRE1α聚集和应激颗粒形成提供了重要的视角,可能对研究细胞应激反应机制和相关疾病治疗具有重要意义。
研究要点
01
肌醇需要酶1(IRE1)是位于内质网上的跨膜蛋白,IRE1是从酵母到哺乳动物保存的最古老的内质网应激传感器,具有一个N端内质网管结构域(LD),用于检测未折叠/错误折叠的蛋白质负载,一个跨膜段,一个细胞质连接体和一个C端蛋白丝氨酸/苏氨酸激酶和核糖核酸内切酶(RNase)结构域,通过激活其胞内段蛋白激酶和核糖核酸内切酶的双重活性,起始并控制着未折叠蛋白响应中最为保守的一条信号通路。
02
在内质网应激条件下,哺乳动物细胞中IRE1α通过剪切编码X盒结合蛋白1(XBP1)mRNA底物产生剪接形式的XBP1s (spliced XBP1)mRNA并翻译出具有转录活性的XBP1s转录因子,以此调控下游应激响应蛋白的表达。大量研究已经证实IRE1α-XBP1通路在细胞命运决定以及包括免疫、代谢和恶性肿瘤在内的许多生物学过程中起关键作用。
03
研究通过免疫荧光检测、活细胞成像、光遗传学、免疫共沉淀及细胞组分分离等实验发现,在哺乳动物细胞中,IRE1α聚集体的形成是与应激颗粒的组装相耦合的内质网膜结合相分离事件,位于内质网的IRE1α聚集簇可以动态地与应激颗粒结合,并与应激颗粒的组装同步偶联,形成IRE1α-SG共聚集簇。
04
IRE1α的细胞质连接部分(连接内质网跨膜结构域和激酶结构域的连接区(Linker))具有固有无序区域(IDR),这是与应激颗粒共凝聚的关键所在。
05
应激颗粒组装的破坏会阻止IRE1α聚集簇的形成,并影响XBP1 mRNA的剪接加工。
06
在内质网应激状况下,IRE1α-SG聚集簇能够富集IRE1α-XBP1信号通路的关键组分:RtcB连接酶和XBP1 mRNA底物。
07
IRE1α-SG聚集簇在时空上的动态特征,表明通过相分离形成的IRE1α-SG聚集簇能够产生更为高效的IRE1α内切酶加工机器,从而增强细胞应对内质网应激的能力,维护细胞的功能稳态。
研究方法
01
IRE1α聚集簇在不同应力下与应激颗粒(SG)共定位
为了探讨不同应激条件下,IRE1α聚集与应激颗粒形成之间的关系,使用化学内质网应激剂thapsigargin (Tg)(56306ES)处理哺乳动物细胞,来测试内源性IRE1α是否能在细胞中形成大的聚集体。使用共聚焦显微镜分析和免疫荧光染色,发现在ER应激的HeLa细胞中,IRE1α形成了大量0.5–5 μm的大聚集体/簇或焦点。结果显示,含有IRE1α聚集簇的细胞百分比随着Tg(56306ES)处理剂量的增加和时间的延长而增加,且在Tg处理后3小时达到高峰。这一发现表明IRE1α聚集簇的形成具有剂量响应性和时间依赖性。(见下图2)
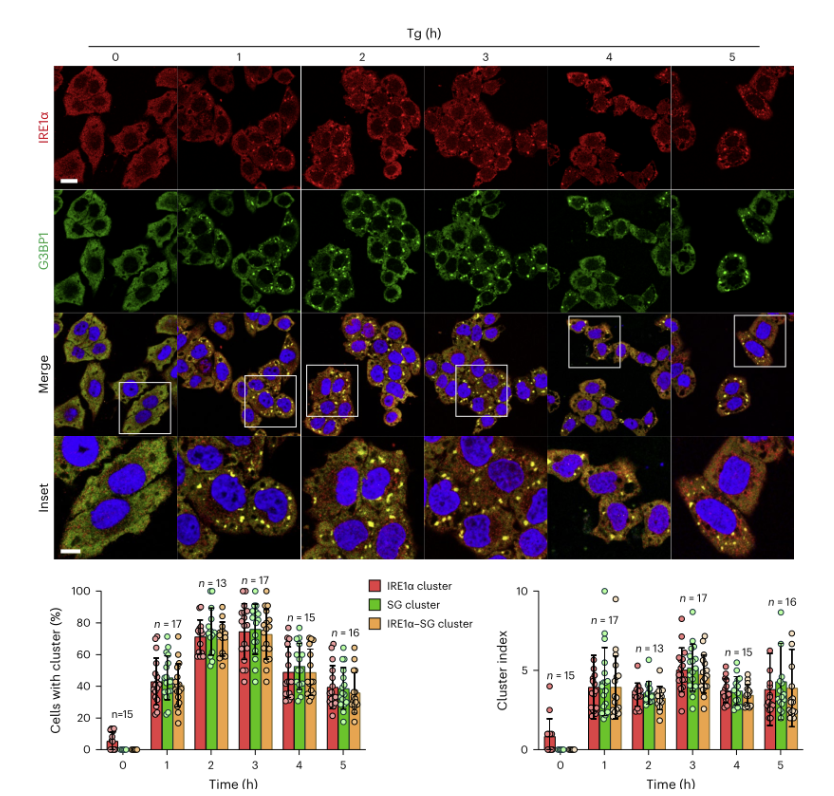
图2. IRE1α簇与SG共定位以响应不同类型的应力
通过对应激颗粒骨架蛋白G3BP1进行共染色,发现应激颗粒的形成也非常强烈,并且与IRE1α聚集体几乎完全共定位。这表明在ER应激条件下,IRE1α聚集簇和应激颗粒之间存在着密切的空间关联。(下图3)
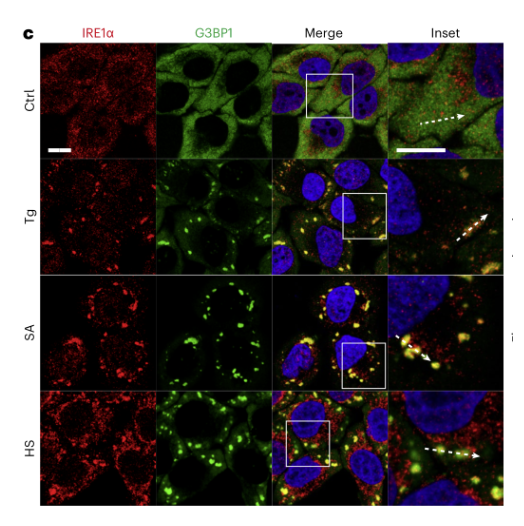
图3. 共焦显微镜图像
由于应激颗粒可以由不同的应激条件诱导,探究IRE1α是否也能响应其他应激因子与应激颗粒共聚集。结果表明,与thapsigargin (Tg)诱导的ER应激相比,使用硫代砷酸钠(SA)和热应激(HS)处理能更有效地诱导大型IRE1α聚集簇的形成。这些聚集簇主要与G3BP1阳性的应激颗粒共定位。(下图4)
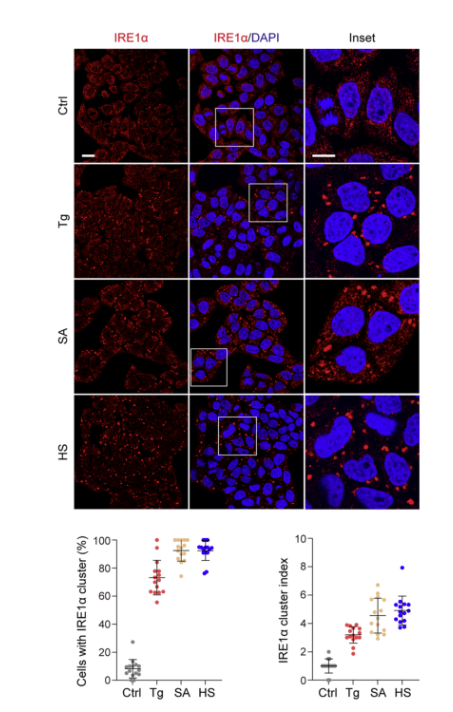
图4. 内源性IRE1α在不同类型的应激反应中形成大簇
线性分析IRE1α/G3BP1双阳性聚集体的结果也表明了这种共定位现象。同时,在使用TIA1(另一个SG支架标记)共染色时,也在HeLa细胞中检测到共定位的IRE1α–SG聚集体的形成。(下图5)
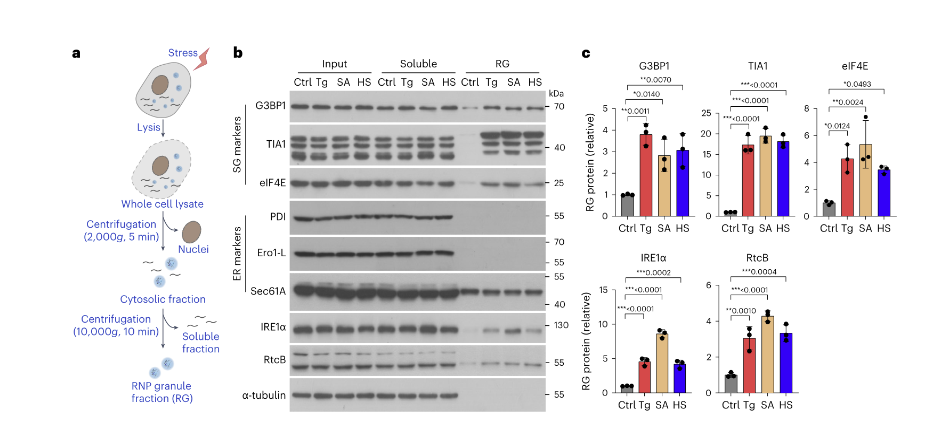
图5. IRE1α蛋白与SG组分的增强结合
研究表明,大型IRE1α聚集体的组装与SG形成有选择性的关联,这是一个普遍的、与细胞类型无关的事件,而不是特定于ER应激的现象。接下来探究生理相关性的应激条件,如缺氧和代谢紊乱对聚集体形成的影响。虽然,在暴露于缺氧条件(1% O2)的HeLa细胞中,没有观察到大型IRE1α聚集体或SGs的形成。但是,在葡萄糖剥夺条件下,检测到了共定位的IRE1α–SG聚集体,特别是在存在糖酵解阻断剂2-脱氧-D-葡萄糖(2-DG)(54104ES)的情况下,这种现象尤为强烈,而2-DG并不引起eIF2α的磷酸化。由于2-DG抑制己糖激酶,从而影响己糖生物合成途径,可能会阻断ER蛋白的糖基化,这些数据表明IRE1α–SG的共聚集也是对营养/代谢应激的响应行为,可能源于ER蛋白折叠的扰动。(下图6)
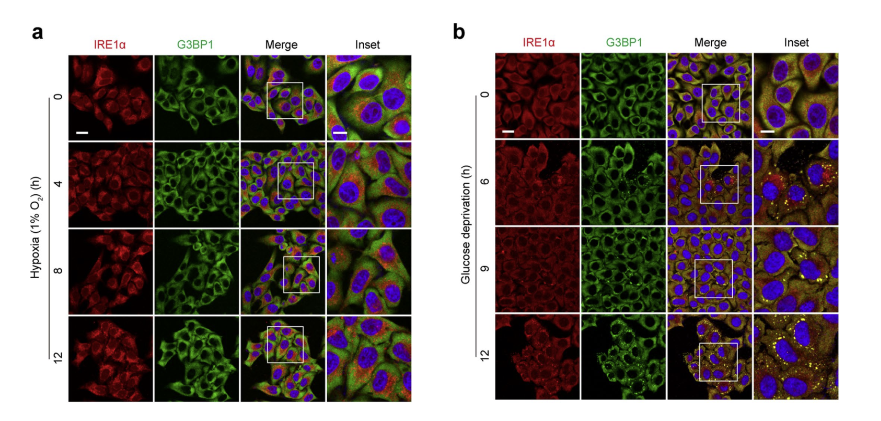
图6. 缺氧和葡萄糖剥夺反应中IRE1α-SG簇形成的分析
02
IRE1α在ER处动态地与SG相连
接下来,团队研究了应激HeLa细胞中IRE1α–SG簇的动力学和结构特征。在消除应激源后,可见IRE1α簇与SG协同减少。表明在应激恢复过程中,IRE1α聚集簇也与SG分解动态耦合。此外,使用增强型绿色荧光蛋白(EGFP)-Sec61β对ER网络进行可视化显示,利用二聚化依赖性荧光蛋白(ddFP)报告系统监测来自不同二聚化伴侣的荧光缩合信号,可见IRE1α/G3BP1双阳性簇围绕ER小管分布在整个细胞质中,IRE1α与核心SG支架蛋白的结构相互作用。(下图7)
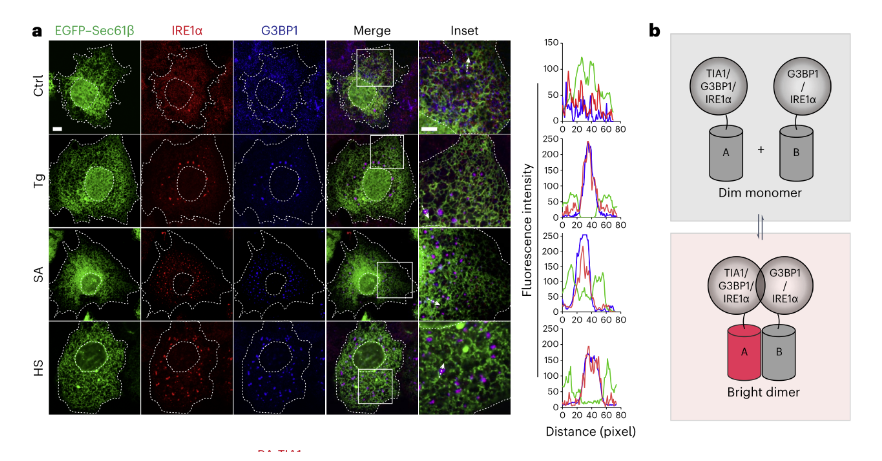
图7. ER处IRE1α-SG团簇的动力学特征
COS7细胞的共聚焦显微镜图像显示,内质网应激诱导IRE1α-YFP与G3BP1/TIA1阳性SG的各种合并或并置聚集,其中一些反映了内质网处缩合物的不对称分层结构。此外,在Tg和SA应激的细胞中,IRE1α/G3BP1聚集与IRE1α/TIA1聚集基本重叠,表明IRE1α与SG支架蛋白的复杂相互作用。实验还观察到聚结的IRE1α/G3BP1颗粒在Tg诱导的内质网应激过程中在ERin细胞处积极进行分裂和融合。这些结果表明,IRE1α聚集簇作为其整体伙伴与SG动态相连。(见下图8)
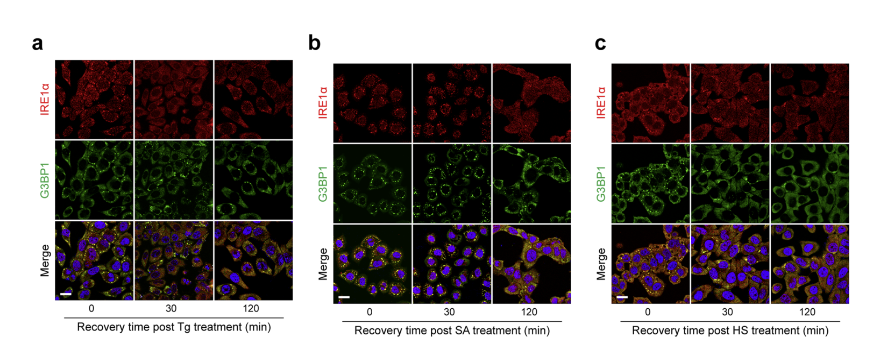
图8. 应力恢复过程中聚结IRE1α-SG团簇的协同还原
03
IRE1α的胞质连接子对与SGs的共聚集至关重要
考虑到SGs的胞质位置,我们使用其细胞质部分的缺失突变体确定了IRE1α与SGs相关的结构域结构。共免疫沉淀分析表明,在内质网应激下,IRE1α_ΔL蛋白的胞浆连接区缺失显著削弱了其与SG蛋白的结合,而IRE1α-ΔR或IRE1α/ΔKR蛋白的胞质连接区缺失则显著削弱了与SG蛋白之间的结合。研究表明,IRE1α的胞质连接子对其与SGs的结合至关重要。IRE1α的细胞质连接内质网跨膜结构域和激酶结构域的连接区(Linker))具有固有无序区域(IDR),这是推进其与应激颗粒共聚集的关键结构域。
04
SG聚结控制IRE1α的聚集和活性
接下来,研究探讨IRE1α聚集是否会影响SG的形成,反之亦然。在不同的应力下,IRE1α的损失不影响IRE1α耗尽的HeLa细胞中SG的形成。此外,对G3BP1颗粒与IRE1α缺陷细胞中的ER膜结合的钙结合蛋白(CalN)的相关性的ddFP报告子分析显示,在ER应激下,SG–ER接触与对照细胞相当。因此,IRE1α聚集不会严重影响SG形成的细胞能力,也不是将SG与ER微管连接的关键因素,但SG组装的破坏会阻止IRE1α聚集簇的形成,并对XBP1 mRNA的剪接加工效率造成影响。
05
SG凝聚促成了更高效的IRE1α加工机器
最后研究探讨了较高IRE1α RNA酶输出的分子基础,即XBP1 mRNA剪接,这是由内质网应激下形成的大IRE1α-SG簇引起的。实验通过检测IRE1α-相邻蛋白质中IRE1α–XBP1轴的关键成分的变化,发现在内质网应激状况下,IRE1α-SG聚集簇能够富集IRE1α-XBP1信号途径中的关键成分:RtcB连接酶和XBP1 mRNA底物。相分离形成的IRE1α-SG聚集簇能够产生更为高效的应激处理工作站——IRE1α内切酶加工站,增强细胞的应激能力,高效维护细胞稳态。
翌圣助力产品
在该研究中,研究团队使用了翌圣生物G418 Sulfate(Geneticin) 遗传霉素(60220ES)进行细胞转染和筛选:

使用Typan Blue Staining Cell Viability Assay Kit台盼蓝染色法细胞活力检测试剂盒(40208ES)进行细胞活力检测:
产品推荐
|
产品名称 |
货号 |
规格 |
|
60220ES03 |
1g |
|
|
60220ES08 |
5g |
|
|
Typan Blue Staining Cell Viability Assay Kit 台盼蓝染色法细胞活力检测试剂盒 |
40208ES60 |
100T |
|
40208ES76 |
500T |
